- Anúncios -
O que significa toy?
Já se perguntou o que significa toy? A palavra é de origem inglesa e se refere a um brinquedo ou objeto de coleção. Descubra mais sobre esse termo e como ele vem sendo utilizado no Brasil.
O que significa central?
Central é uma palavra que carrega consigo uma essência poderosa de conexão e controle. Seja no contexto geográfico, onde representa o ponto de convergência, ou no sentido figurado, onde alude ao poder de comando. Explorar o significado dessa palavra multifacetada nos leva a refletir sobre o poder do centro.
O que significa mere?
Mere. Três letras, uma infinidade de significados. Essa pequena palavra da língua portuguesa carrega consigo um mundo de possibilidades. Tão simples, porém tão complexa. Mas afinal, o que significa "mere"? Vamos explorar as camadas dessa palavra intrigante e desvendar seus segredos. Descubra se você merece o conhecimento que está por vir!
Medicina
O que significa anemia?
Anemia - a palavra que ecoa delicadamente no universo da saúde. Mas,…

Create an Amazing Newspaper
Siga-nos
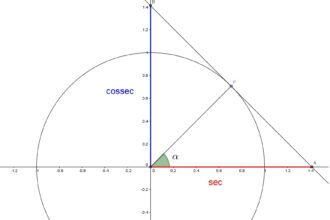
O que significa trigonometria na matemática?
O que é a trigonometria? Para alguns, é o estudo dos números…
O que significa axioma na matemática?
Você já se perguntou o que significa axioma na matemática? Bem, prepare-se…
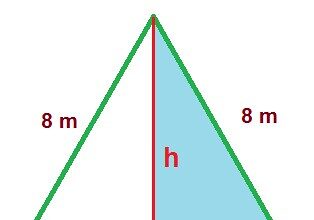
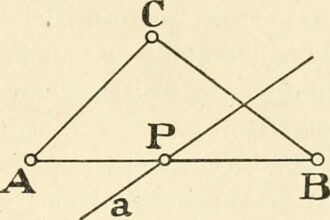
O que significa isósceles?
O que significa isósceles? Essa palavra enigmática carrega consigo mistérios geométricos e…
O que significa subtração?
A subtração é um conceito matemático que pode parecer complicado à primeira…
O que significa aproximação na matemática?
A aproximação na matemática é uma poderosa ferramenta que nos permite obter…
O que significa geometria?
Geometria, a palavra que une formas, linhas e figuras num mundo de…
O que significa arco na matemática?
Na matemática, o arco é um elemento fundamental que nos leva a…
O que significa antilogaritmo na matemática?
Antilogaritmo, uma palavra intrigante no mundo da matemática. Mas o que será…

Create an Amazing Newspaper
Conteúdo patrocinado
O que significa capitalismo?
O que significa capitalismo? É um conceito que se desdobra em um verdadeiro oceano de interpretações e debates. Desde sua origem na Revolução Industrial até os dias atuais, o capitalismo abrange os pilares da propriedade privada, da livre iniciativa e do mercado competitivo. Mas, será que esse sistema econômico tem se mostrado eficiente e sustentável para todos? Exploraremos essas questões e muito mais neste artigo, mergulhando nas profundezas desse complexo sistema que molda nossa sociedade contemporânea.
O que significa ASAP?
O que significa ASAP? É uma sigla amplamente utilizada em todo o mundo dos negócios, que expressa a urgência e importância de uma ação imediata. Uma expressão curta, mas poderosa,…
Top Autores
Stay Up to Date
Subscribe to our newsletter to get our newest articles instantly!