- Anúncios -
O que significa extension?
O que significa extension? Uma palavra curta, mas com um universo de significados. É como um fio que se estende, expandindo possibilidades e conexões. Uma extensão pode ser um prolongamento físico ou uma ampliação conceptual. É aí que reside a magia dessa palavra, na sua capacidade de transcender limites. Extension é o elo que une o presente ao futuro, o…
O que significa lot?
O significado de "lot" é algo que ultrapassa a mera definição. É o pulsar da vida em seus múltiplos significados. Do fervilhar da agitação nas ruas às conexões que se estabelecem entre pessoas, "lot" é a essência de um encontro, de uma experiência compartilhada. Renda-se ao ritmo frenético do "lot" e permita-se viver intensamente cada momento.
O que significa squeeze?
Imagine-se em meio a um encontro animado, quando de repente alguém solta a pergunta enigmática: "O que significa squeeze?" Talvez esse termo seja desconhecido para muitos, mas a resposta está ao alcance de suas mãos. Descubra agora mesmo o significado desse termo curioso e amplie seu vocabulário. Leia mais!
Medicina
O que significa infarto do miocárdio?
Você já tentou ouvir o seu coração? O som que ecoa dentro…

Create an Amazing Newspaper
Siga-nos
O que significa cálculo?
Cálculo, um termo que provoca curiosidade e talvez até um certo temor…
O que significa algoritmo na matemática?
Algoritmo na matemática é como uma incrível coreografia numérica, uma dança entre…
O que significa potenciação?
Você provavelmente já se deparou com a potenciação em algum momento da…
O que significa dividendo?
Você já se perguntou o que significa dividendo? Essa é uma palavrinha…
O que significa denominador?
Você já se perguntou o que significa denominador? Uma palavra tão peculiar…
O que significa área na matemática?
Na matemática, a área é um conceito fundamental que nos permite medir…
O que significa quociente?
O que significa quociente? Uma pergunta que pode desencadear diversos pensamentos profundos…
O que significa tangente na matemática?
A tangente, em sua essência matemática, é uma relação misteriosa entre duas…

Create an Amazing Newspaper
Conteúdo patrocinado
O que significa capitalismo?
O que significa capitalismo? É um conceito que se desdobra em um verdadeiro oceano de interpretações e debates. Desde sua origem na Revolução Industrial até os dias atuais, o capitalismo abrange os pilares da propriedade privada, da livre iniciativa e do mercado competitivo. Mas, será que esse sistema econômico tem se mostrado eficiente e sustentável para todos? Exploraremos essas questões e muito mais neste artigo, mergulhando nas profundezas desse complexo sistema que molda nossa sociedade contemporânea.
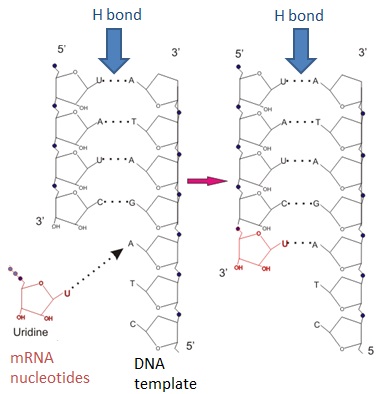
O que significa RNA?
Se você olhar para a própria essência da vida, vai se deparar com o RNA. Essas três letras representam uma das moléculas mais fundamentais e intrigantes da biologia. Mas o…
Top Autores
Stay Up to Date
Subscribe to our newsletter to get our newest articles instantly!